Team:KIT-Kyoto/Notebook-week3
From 2010.igem.org
 
|
Photograph
August 22
Time
- 9:00~
Member
- 古道、臼井、中村、足立
- Furumichi,Usui,Nakamura,Adachi
Title
- (1) PCRで複製したAcrAB,oxyRの電気泳動 [足立、臼井]
- (2) pSB4A5(K193602),pSB1AK3(I732950)のプレカルチャー [臼井]
- (3) AcrAB,oxyRのPCR [臼井]
Purpose
- Agarose gel electrophoresis of AcrAB and OxyR.[Adachi,Usui]
- Pre-culture of pSB4A5(K193602),pSB1AK3(I732019,I732950).[Usui]
- PCR of AcrAB and OxyR.[Usui]
- (1) PCRによってAcrAB,oxyRの大量複製に成功しているかのチェック
- (2) pSB4A5(K193602),pSB1AK3(I732019,I732950)の培養
- (3) AcrAB,oxyRのPCR
Method
- (1) PCRで複製したAcrAB,oxyRの電気泳動
- 8月21日(土)にPCRで複製したAcrAB(DH5α)AcrAB(JM109),oxyR(DH5α),oxyR(JM109)を
- 各4本ずつ計12本電気泳動を行うことによりPCR産物の複製の程度を調べた
- AcrAB(DH5α)AcrAB(JM109),oxyR(DH5α),oxyR(JM109)を
- 各サンプルそれぞれ15μLずつにLoading Buffer 1μLを加えコームに流した
- ↓1kbp radderをマーカーとして使用し100vで20分間電気泳動を行った
- ↓EtBr(10mg/ml)で20分間染色した
- ↓泳動結果を写真撮影した
- ※結果は写真参照
- (2) pSB4A5(K193602)[MelA],pSB1AK3(I732950)[lacZ]のプレカルチャー
- プレート培地上からpSB4A5(K193602),pSB1AK3(I732950)のシングルコロニーをピックアップした
- ↓各2本ずつ、3mLの液体LB培地(+amp)を使用し、37℃で振とう培養した(overnight)
- (3) AcrAB,oxyRのPCR
- AcrAB,oxyRに関して8月21日の実験結果より
- 以下のようにMg4μL、テンプレートDNA1μLの試薬組成でAcrAB,oxyRのプロモーターを大量に複製した
<PCR試薬組成> PrimerF 1.5μL PrimerR 1.5μL dNTPs 5μL KOD-Plus- Buffer 5μL テンプレートDNA
(DH5α,JM109)1μL MgSO4 4μL H2O 31μL KOD-Plus- 1μL total 50μL <設定> Pre Denature Denature Annealing Extention +Extention 94℃ 94℃ 62.8℃ 68℃ 68℃ 4℃ 2min 15sec 30sec 20sec 2min 保持 35サイクル
Results
- (1) 8月21日(土)のPCR試薬組成(2)で電気泳動をしたところ、
- AcrAB(DH5α)
- AcrAB(JM109)
- oxyR(DH5α)
- oxyR(JM109)
- それぞれでバンドが確認できなかった
- (2) 翌日pSB4A5(K193602),pSB1AK3(I732019,I732950)は培養が成功していたので、
- 後日使うまで、冷蔵保存しておいた
- (3) 詳しい結果は23日(2)に掲載した
August 23
Time
- 9:00~21:00
Member
- 岩城,竹内,中村,臼井,吉村
- Iwaki,Takeuchi,Nakamura,Usui,Yoshimura
Title
- (1) 大腸菌の生存曲線を調べる [吉村]
- (2) dps,grxA,trxCおよびoxyRのPCR+電気泳動によるチェック [中村、臼井]
- (3) dps,acrABのDNA濃度測定 [中村、臼井]
Purpose
- The survivorship curve of E. coli is examined. [Yoshimura]
- PCR of dps,AcrAB,trxC,and OxyR.And Comfirm those by agarose gel electrophoresis.[Nakamura,Usui]
- Measurement of density of DNA of dps and acrAB.[Nakamura,Usui]
- (1) 大腸菌の生存曲線を調べる
- (2) 活性酸素応答のプロモーターをPCRで増やし、ちゃんと増えたかを泳動でチェック
- (3) dps,acrABのDNA濃度を測定し、PCRして増やすのに十分な濃度であるかを確認する。
Method
- (1) 大腸菌の生存曲線を調べる
- 30% H2O2(500μl)とミリQ(600μl)で4MのH2O2をつくった
- ↓4M H2O2(250μl)とミリQ(750μl)で1MのH2O2をつくった
- ↓以下10倍希釈していき、1nM H2O2まで作成した
- 次にDH5αのシングルコロニーをピックアップした
- ↓LB(-amp)2mlでプレカルチャーを行った
- ↓(37℃でインキュベート、overnight)
- (2) dps,grxA,trxCおよびoxyRのPCR+電気泳動によるチェック
- dps,grxA,trxCおよびoxyRのPCRを以下の組成、設定で行った
<組成> (dps,grxA,trxC) (oxyR) PrimerR 1.5μL PrimerR 3μL PrimerF 1.5μL PrimerF 3μL dNTP 5μL dNTP 20μL Buffer 5μL Buffer(KOD-FX) 50μL DNA(DH5α) 0.5μL DNA(DH5α) 2μL MgSO4 4μL MilliQ 20μL MilliQ 31.5μL KOD-FX 2μL KOD-Plus 1μL 全量 50μL 全量 100μL <PCRの設定> (dps,grxA ,trxC) (oxyR) 1.Pre Penature 94℃―2min 94℃―2min 2.Denature 94℃―15sec 94℃―10sec 3.Annealing 59.7℃―30sec 52℃―30sec 4.Extension 68℃―30sec 68℃―45sec 5.+Extension 68℃ー2min 68℃ー2min
またPCR後、それぞれ電気泳動によるチェックを以下の条件で行った
1回目 100V、20min EtBr 20min
2回目 135V、15min EtBr 10min
- (3) dps,acrABのDNA濃度測定
- 吸光計により、dpsとacrABのOD260を5回測定し、その平均をとった
Results
- (1) 実験結果は25日のノートに記載した
- (2) 1回目の電気泳動で、grxA、trxC のバンドがほとんど確認されなかった
- 2回目を行ったところ、1回目よりはバンドがよく見えたが、十分に増えたとは言えなかった
- *詳細は写真参照
- (3) 吸光度の測定結果
- 以下の吸光度の結果から、PCRが十分と判断された
吸光度 dps acrAB 1回目 0.340 0.565 2回目 0.329 0.555 3回目 0.314 0.556 4回目 0.309 0.556 5回目 0.312 0.551 平均値 0.321 0.557
August 24
Time
- 9:00~21:00
Member
- 岩城,竹内,中村,臼井,吉村
- Iwaki,Takeuchi,Nakamura,Usui,Yoshimura
Purpose
- The survivorship curve of E. coli is examined. [Yoshimura]
- Confirmation of PCR products(dps and AcrAB) by Agarose gel electrophoresis.[Nakamura,Usui]
- Dps and AcrAB digestion by restriction enzymes.[Nakamura,Usui]
- (1) 大腸菌の生存曲線を調べる(2日目) [吉村]
- (2) dps、acrABの電気泳動によるPCRの確認 [中村、臼井]
- (3) dps、acrABの制限酵素処理 [中村、臼井]
Method
- (1) 大腸菌の生存曲線を調べる(2日目)
- 前日にプレカルチャーしたLB 2 mlにLB(-amp)(2ml)を加え濃度調整、濁度を計った
- ↓O.D.600=0.4~0.6まで培養することを目的とした
- 10:00 O.D.600=0.310
- 11:30 O.D.600=0.908 (濁度が高すぎたため、この溶液5mlをLB20mlに植え継ぎ培養しなおした)
- 11:50 O.D.600=0.152
- 13:00 O.D.600=0.502 (目的の濃度に達したため、培養を停止した)
- ↓培養後、2mlずつ分注し、それぞれにH2O2を加えた
- ↓加えた後、37℃で1時間培養した
- ↓培養後、LB1mlに培養液をそれぞれ1μlずつ分注し、その内10μlをLBプレートにまいた
- ↓37℃でインキュベートを行った(overnight)
- (2) dps、acrABの電気泳動
- 前日にPCRしておいたdps、acrAB(50μL×2本ずつ)を電気泳動し、EtBr処理の後、写真を撮影した
- その条件を以下に示す
- <条件>
- DNA 10μL
- 1%アガロースゲルで135V、15min
- EtBr処理 10min
- (3) dps、acrABの制限酵素処理
- PCR後のdps、acrABをライゲーション用に制限酵素処理を行った
- 制限酵素処理の組成を以下に示す
- <組成>
- DNAsol 40μL
- 1×H.Buffer 5μL
- EcoRⅠ 1μL
- SpeⅠ 1μL
- H2O 3μL
- これを、37℃で3.5hインキュベートした後、冷蔵保存した
Results
- (1) 実験結果は25日に記載
- (2) 薄いバンドが目的の位置に確認された
- *詳細は写真参照
- (3) 制限酵素処理済のdps、acrABは25日にライゲーションを行う予定
August 25
Time
- 9:00~21:00
Member
- 岩城,古道,竹内,吉村
- Iwaki,Furumichi,Takeuchi,Yoshimura
Purpose
- The survivorship curve of E. coli is examined. [Yoshimura]
- The survivorship curve of E. coli is examined. [Yoshimura]
- Pre-culture of pSB1A2(I13507) , pSB1A2(E0430) and pSB1A2(J22005).[Furumichi]
- Transform pSB1A2(I13507) and pSB1A2(E0420) into the competent cells.[Furumichi]
- Ligation of AcrAB-pSB6A1(K121013) and dps-pSB6A1(K121013).[Iwaki,Takeuchi]
- DNA miniprep pSB3K3(J04450),pSB6A1(K121013),pSB1A2(E0240),pSB1AK3(I732950) and pSB4A5(K193602).[Iwaki,Takeuchi]
- (1) 大腸菌の生存曲線を調べる(3日目) [吉村]
- (2) 大腸菌の生存曲線を調べる(1日目) [吉村]
- (3) pSB1A2(I13507),pSB1A2(E0430),pSB1A2(J22005)のプレカルチャー [古道]
- (4) pSB1A2(I13507),pSB1A2(E0420)をDH5αに形質転換 [古道]
- (5) dps,AcrAB-pSB6A1(K121013)のLigation [岩城、竹内]
- (6) pSB3K3(J04450),pSB6A1(K121013),pSB1A2(E0240),
- pSB1AK3(I732950),pSB4A5(K193602)のプラスミド抽出 [岩城、竹内]
Method
- (1) 大腸菌の生存曲線を調べる
- プレートにコロニーが生えているか確認し、コロニーの数を数えた
- (2) 過酸化水素の浸透性を利用し、大腸菌の生存曲線を調べる
- 実験(1)でcontrolのプレートにコロニーが生えなかったので、違った方法でプレートを作成した
- DH5αをO.D.600=0.4~0.6になるまで培養した
- ↓培養後、LB1mlに対して、培養液を1ml加えて希釈した
- ↓希釈した培養液10μlをLBプレートにまき、37℃で1時間インキュベートした
- ↓1時間後、過酸化水素水をLBプレートの中心に20μl滴下し、
- 滴下した過酸化水素水がLBプレートに完全に浸透するまで待った
- ↓完全に浸透したのを確認し、37℃でインキュベートした(overnight)
- (3) pSB1A2(I13507),pSB1A2(E0430),pSB1A2(J22005)のプレカルチャー
- pSB1A2(I13507),pSB1A2(E0430),pSB1A2(J22005)の
- コロニーをピックアップしLB液体培地(+amp)に入れた
- ↓37℃で振とう培養した
- (4) pSB1A2(I13507),pSB1A2(E0420),をDH5αに形質転換
- pSB1A2(I13507),pSB1A2(E0420)をそれぞれ1µl取り、そこにDH5α 100µlを加え混ぜた
- ↓氷上、30min
- ↓42℃で45秒間、熱ショックを与えた
- ↓氷上、2min
- ↓SOC培地0.9mlを加えた
- ↓37℃で1時間振とう培養した
- ↓400µlずつLB(+amp)プレートに播いて37℃でインキュベートした(overnight)
- (5) dps,AcrAB-pSB6A1(K121013)のLigation
EcoRⅠ-SpeⅠで制限酵素処理したdps,AcrABと
↓ゲルのレーンには左2列にpSB6A1(K121013)を、右2列にdpsを流した
EcoRⅠ-XbaⅠで制限酵素処理したpSB6A1(K121013)に電気泳動を行い、
ゲル抽出、ライゲーションを行った
dpsは前日(24日)に制限酵素処理したものを用いた
↓pSB6A1(K121013)の制限酵素処理を行った
↓右表に従い試薬を混合し、37℃で30分間インキュベートした
↓CIAP 1μLを加えて、37℃で30分間インキュベートした
↓dpsとpSB6A1(K121013)それぞれにOrange Loading Dyeを10μLずつ加え、
30μずつ、レーンにアプライした
<試薬組成> DNAsol 30μL M buffer 5μL EcoRⅠ 1μL XbaⅠ 1μL H2O 13μL 全量 50μL
- (6) 大量培養後のアルカリミニプレップ
- 以下の5種類のベクターを用意した
- pSB3K3(J04450)
- pSB6A1(K121013)
- pSB1A2(E0240)
- pSB1AK3(I732950)
- pSB4A5(K193602)
- 菌液をチューブに入る量だけ入れた
- ↓4℃、14,000rpmで5分間遠心した
- ↓上清を捨てた後、菌液を更に追加した
- ↓4℃、14,000rpmで5分間遠心した
- ↓以上の操作を菌液がなくなるまで繰り返した
- ↓solⅠ 4mLを加え、vortexした
- ↓solⅡ 200μLを加えon ice 5min.
- ↓solⅢを150μL加えon ice 5min.
- ↓4℃、14,000rpmで10分間遠心した
- ↓イソプロパノール400μL(等量)を加えた
- ↓4℃、14,000rpmで20分間遠心した
- ↓70%EtOHを200μL加えた
- ↓4℃、14,000rpmで10分間遠心した
- ↓遠心乾燥を行った
- ↓これをTE(RNase)30μLに溶解し、冷蔵保存した
- (1) コロニー数を数えて以下の表にまとめた
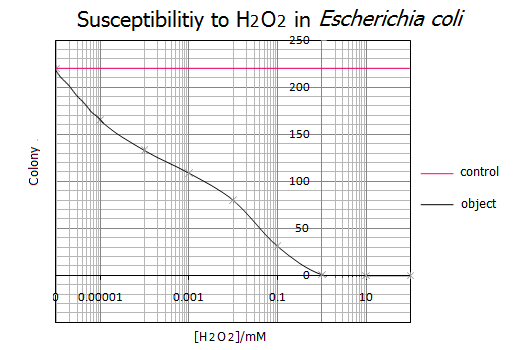
H2O2濃度 0mM(control) 100nM 10nM 1nM 100pM 10pM 1pM コロニー数 0 7 147 59 0 144 289 - controlのプレートになぜかコロニーが生えなかった
- この実験では大腸菌をプレートにまく作業を7回繰り返したため、時間差が生じてしまったのが原因とみられる
- とにかくcontrolプレートに大腸菌が生えなければどうしようもないので、この結果は不採用とした
- (2) 8月26日に結果を記載した
- (3) 三本ともプレカルチャーが成功したため、8月27日の実験で使用した
- (4) pSB1A2(I13507) ,pSB1A2(E0420)のトランスフォーメーションは成功した
- よって、8月27日の実験で使用することになった
- (5) ゲル抽出に移る前にdpsのバンドが十分量検出できなかったため、
- ゲル抽出、およびLigationを行うことができなかった
- pSB6A1(K121013)のバンドははっきりと見えたが、濃度としては不十分だった
- (6) すべてのプラスミドの濃度が低かった
August 26
Time
- 9:00~
Member
- 岩城,竹内,中村,臼井,足立
- Iwaki,Takeuchi,Nakamura,Usui,Adachi
Purpose
- The survivorship curve of E. coli is examined.[Adachi]
- pSB1A2(E0430),pSB1A2(J22005) and pSB1A2(I13507) digestion by restriction enzymes.And confirm by Agarose gel electrophoresis.[Usui]
- PCR of SodA.[Usui]
- (1) 大腸菌の生存曲線を調べ(2日目)、生存曲線を作成 [足立]
- (2) 前日にアルカリミニプレップした
- pSB1A2(E0430),pSB1A2(J22005),pSB1A2(I13507)を制限酵素で処理、処理の確認(電気泳動) [臼井]
- (3) SodAのプロモーターをPCRによって増幅 [臼井]
Method
- (1) 大腸菌の生存曲線を調べる
- 前日まいたコロニーの数を数え、今までのデータを統計、生存曲線を作成した
- (2) pSB1A2(E0430),pSB1A2(J22005),pSB1A2(I13507)の制限酵素処理
右表の組成で制限酵素処理を行い、37℃で1時間インキュベートした
↓結果を写真撮影した
↓電気泳動を以下の方法で行った
1×ladderをマーカーとし、一番左のコームに流した
↓Loading Buffer1μLに対し
pSB1A2(E0430),pSB1A2(J22005),pSB1A2(I13507)をぞれぞれ5μLの割合で加え左から
2∼4番目にpSB1A2(E0430)、
5~7番目にpSB1A2(J22005)、
8番目(右端)にpSB1A2(I13507)をそれぞれコームに流した
<組成> EcoRⅠ lμL PstⅠ 1μL プラスミドDNA 5μL 10×H Buffer 2μL H2O 11μL 全量 20μL
- (3) SodAのPCR処理
下表の組成でテンプレートDNAにDH5αを使用したものを2本(DH5α-1,DH5α-2)、
JM109を使用したものを2本(JM109-1,JM109-2)作製した
↓以下のプログラムでPCR処理を行った<PCR試薬の組成> SodA PrimerF 1.5μL PrimerR 1.5μL dNTPs 5μL KOD-Plus- Buffer 5μL テンプレートDNA
(DH5α,JM109)0.5μL MgSO4 4μL H2O 31.5μL KOD-Plus- 1μL total 50μL
Pre Denature Denature Annealing Extention +Extention 94℃ 94℃ 62.2℃ 68℃ 68℃ 4℃ 2min 15sec 30sec 1min 2min 保持 30サイクル
- (1) 計測した結果を以下にまとめた
[H2O2] 0mM 1nM 10nM 100nM 1μM 10μM 100μM 1mM 10mM 100mM コロニー数 231 220 165 133 109 80 31 1 0 0
- 以下の引用文献を参考に、得られた結果は正しいと判断した
- (2) pSB1A2(E0430)はすべてバンドが見えたので、
- バンドの一番濃かった左から4番目のものを3mLのLB培地(amp+)で37℃で振とう培養した(overnight)
- pSB1A2(J22005)は左から6番目と7番目がバンドが見えたので、
- 7番目のものを3mLのLB培地(amp+)で37℃で振とう培養した(overnight)
- RFP(I13507)はバンドが現れなかった
- (3) PCR処理を行った4つ全てについてSodAが十分量含まれていることが27日(金)の電気泳動によりわかった
August 27
Time
- 9:00~
Member
- 福山,竹内,臼井
- Fukuyama,Takeuchi,Usui
Purpose
- DNA miniprep of psB6A1(K121013).[Takeuchi]
- Making of EtBr.[Takeuchi]
- (1) psB6A1(K121013)のミニプレ [竹内]
- (2) エチジウムブロマイドの作成 [竹内]
Method
- (1) psB6A1(K121013)のミニプレ
- psB6A1(K121013)24mL分を分注した
- ↓遠心1分(20℃、14500rpm)して集菌し、エッペン2本分にした
- ↓上清を捨てた
- ↓SolutionⅠを250μl加えてピペッティングし、ボルテックスした
- ↓SolutionⅡを250μl加えた
- ↓SolutionⅢを350μl加えた
- ↓遠心5分(20℃、14500rpm)
- ↓上清をカラムに入れた
- ↓遠心30秒(20℃、14500rpm)
- ↓抽出液を捨てた
- ↓SolutionⅣを500μlカラムに加えた
- ↓遠心1分(20℃、14500rpm)
- ↓抽出液を捨てた
- ↓SolutionⅤを700μlカラムに加えた
- ↓遠心30秒(20℃、14500rpm)
- ↓抽出液を捨てた
- ↓遠心30秒(20℃、14500rpm)
- ↓抽出液を捨てた
- ↓滅菌水をカラムに入れた
- ↓遠心30秒(20℃、14500rpm)
- ↓溶出液を回収
- ↓酢酸ナトリウムとイソプロパノールを溶出液量に対して、それぞれ1/10倍、等量加えてまぜた
- ↓遠心10分(4℃、14000rpm)
- ↓上清をすて、70%エタノールを各エッペンに800μlずつ加えた
- ↓遠心5分(4℃、14000rpm)
- ↓上清をすて、遠心乾燥機で乾燥
- ↓MilliQを30μl加えた
- ↓二本分のまとめ、そのうち6μLを電気泳動に使用した
- Loading Buffer 1μLをそれぞれに加えコームに流した
- (マーカーは1kbp radderを使用した)
- ↓100V,30min電気泳動しEtBr(10mg/mL)で染色
- ↓泳動結果を写真で撮影した
- (2) エチジウムブロマイドの作成
- MilliQを40mLに臭化エチジウムブロマイド溶液(0.44mg/mL)を1滴加えた
Results
- (1) 十分に濃いバンドが確認できた
- これより、ミニプレによるプラスミド抽出は成功したと考えられる
- (2) 1滴では十分な染色が行えなかったので、後日さらに3滴付加したところ十分に使用できるようになった
Consideration
- (1) 次回からは100倍希釈による吸光度測定を行い、プラスミド濃度を測るべき
- (2) これより40mL分を作る時は少なくとも4滴は加えた方が使用に十分な染色液が作れると考えられる
August 28
Time
- 9:00~
Member
- 岩城,竹内,中村,臼井,革島
- Iwaki,Takeuchi,Nakamura,Usui,Kawashima
Purpose
- Making of primer mix of AcrAB,dps,SodA and OxyR.[Takeuchi,Usui]
- PCR of AcrAB and OxyR.[Iwaki]
- PCR of dps and hemH.[Usui]
- Agarose gel electrophoresis of dps and hemH.[Iwaki]
- DNA miniprep of pSB1A2(E0420) and pSB1A2(I13522).[Nakamura]
- Agarose gel electrophoresis of pSB1A2(E0420) and pSB1A2(I13522).[Nakamura]
- (1) AcrAB,dps,SodA,OxyRのプライマーミックス(PM)の作成 [竹内、臼井]
- (2) AcrAB,oxyRのPCR [岩城]
- (3) dps,hemHのPCR [臼井]
- (4) dps,hemHの電気泳動 [岩城]
- (5) pSB1A2(E0420)、pSB1A2(I13522)のミニプレ [中村]
- (6) pSB1A2(E0420)、pSB1A2(I13522)の電気泳動 [中村]
Method
- (1) Primer MIXの作成
- AcrAB,dps,SodA,OxyRのPrimerF,PrimerRそれぞれ10μLずつを80μLのH2Oに溶解し、
- 全量100μL、10pmol/μLにした溶液(PrimerMIX)を作成した
- (2) AcrAB,oxyRのPCR
<PCR試薬組成> AcrAB oxyR PrimerF 1.5μL 1.5μL dNTPs 5μL 5μL KOD-Plus- Buffer 5μL 5μL テンプレートDNA(DH5α) 1μL 1μL MgSO4 4μL 4μL H2O 33μL 33μL KOD-Plus- 1μL 1μL total 50μL 50μL <設定> Pre Denature 94℃ 2min 35Cycles↓ Denature 94℃ 15sec Annealing 62.8℃ 30sec Extension 68℃ 30sec +Extension 68℃ 2min 4℃ ∞
- (3) dps,hemHのPCR
<PCR試薬組成> dps hemH PrimerF 1.5μL 1.5μL dNTPs 5μL 5μL KOD-Plus- Buffer 5μL 5μL テンプレートDNA(DH5α) 0.5μL 0.5μL MgSO4 4μL 4μL H2O 33μL 33μL KOD-Plus- 1μL 1μL total 50μL 50μL <設定> Pre Denature 94℃ 2min 35Cycles↓ Denature 94.0℃ 15sec Annealing 60.0℃ 30sec Extension 68.0℃ 30sec +Extension 68.0℃ 2min 4℃ ∞
- (4) dps,hemHの電気泳動
- (3)のPCRの後、増幅を確認するために電気泳動にかけ、写真撮影を行った
- 諸条件を以下に示す
- <条件>
- DNA量 5μL
- 電気泳動 100V 30min
- EtBr処理 20min
- (5) pSB1A2(E0420)、pSB1A2(I13522)のミニプレ
- pSB1A2(E0420)、pSB1A2(I13522)について同様の操作を行った
- 培養後の培養液1.5mLを1.5mLマイクロチューブに移した
- ↓4℃、14,000rpm、5min遠心
- ↓上澄みをデカンテーションで取り除いた
- ↓菌体ペレットを氷冷したSoltionⅠ100μLに懸濁(vortex)
- ↓SolutionⅡ200μLを加え混合
- ↓5min on ice
- ↓SolutionⅢ150μLを加え混合(vortex)
- ↓5min on ice
- ↓4℃、14,000rpm、10min遠心
- ↓上澄みを新しい1.5mLマイクロチューブに移した
- ↓等量(400μL)イソプロピルアルコールを加えた
- ↓4℃、14,000rpm、20min遠心
- ↓70%EtOHを200μL加える
- ↓4℃、14,000rpm、10min遠心
- ↓30sec 遠心乾燥
- ↓TE30μL加えてプラスミドDNAとした
- (6) pSB1A2(E0420)、pSB1A2(I13522)の電気泳動
- pSB1A2(E0420)、pSB1A2(I13522)のミニプレ後、電気泳動を行いチェックした
- <条件>
- DNA量 5μL
- 電気泳動 100V 20min
- EtBr処理 20min
Results
- (1) 今回作成したPMは今後使用していく予定
- (2) 後日電気泳動により増幅の確認を行う
- (3,4) hemH、dpsに関してははっきりとバンドが見え、増幅が確認された
- (5,6) pSB1A2(E0420)、pSB1A2(I13522)共に明瞭なバンドが確認された
- *詳細は写真参照
Remarks
- (3,4)SpeⅠが届き次第、制限酵素処理を行う
- (5,6)pSB1A2(E0420)、pSB1A2(I13522)のプレートからシングルコロニーをピックアップして、LB培地(1)で培養
- (3) dps,acrABのDNA濃度測定
 "
"