Template:KIT-Kyoto-Augsut26
From 2010.igem.org
Revision as of 05:36, 4 October 2010 by Matsubara3 (Talk | contribs)
August 26
Time
- 9:00~
Member
- 岩城,竹内,中村,臼井,足立
- Iwaki,Takeuchi,Nakamura,Usui,Adachi
Purpose
- (1) 大腸菌の生存曲線を調べ(2日目)、生存曲線を作成 [足立]
- (2) 前日にアルカリミニプレップした
- pSB1A2(E0430),pSB1A2(J22005),pSB1A2(I13507)を制限酵素で処理、処理の確認(電気泳動) [臼井]
- (3) SodAのプロモーターをPCRによって増幅 [臼井]
Method
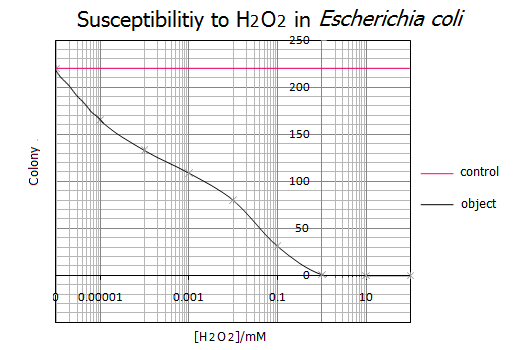
- (1) 大腸菌の生存曲線を調べる
- 前日まいたコロニーの数を数え、今までのデータを統計、生存曲線を作成した
- (2) pSB1A2(E0430),pSB1A2(J22005),pSB1A2(I13507)の制限酵素処理
右表の組成で制限酵素処理を行い、37℃で1時間インキュベートした
↓結果を写真撮影した
↓電気泳動を以下の方法で行った
1×ladderをマーカーとし、一番左のコームに流した
↓Loading Buffer1μLに対し
pSB1A2(E0430),pSB1A2(J22005),pSB1A2(I13507)をぞれぞれ5μLの割合で加え左から
2∼4番目にpSB1A2(E0430)、
5~7番目にpSB1A2(J22005)、
8番目(右端)にpSB1A2(I13507)をそれぞれコームに流した
<組成> EcoRⅠ lμL PstⅠ 1μL プラスミドDNA 5μL 10×H Buffer 2μL H2O 11μL 全量 20μL
- (3) SodAのPCR処理
下表の組成でテンプレートDNAにDH5αを使用したものを2本(DH5α-1,DH5α-2)、
JM109を使用したものを2本(JM109-1,JM109-2)作製した
↓以下のプログラムでPCR処理を行った<PCR試薬の組成> SodA PrimerF 1.5μL PrimerR 1.5μL dNTPs 5μL KOD-Plus- Buffer 5μL テンプレートDNA
(DH5α,JM109)0.5μL MgSO4 4μL H2O 31.5μL KOD-Plus- 1μL total 50μL
Pre Denature Denature Annealing Extention +Extention 94℃ 94℃ 62.2℃ 68℃ 68℃ 4℃ 2min 15sec 30sec 1min 2min 保持 30サイクル
- (1) 計測した結果を以下にまとめた
[H2O2] 0mM 1nM 10nM 100nM 1μM 10μM 100μM 1mM 10mM 100mM コロニー数 231 220 165 133 109 80 31 1 0 0
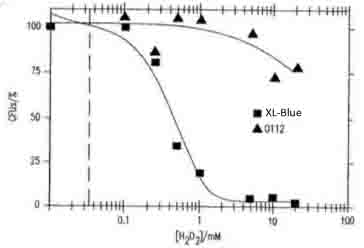
- 以下の引用文献を参考に、得られた結果は正しいと判断した
- (2) pSB1A2(E0430)はすべてバンドが見えたので、
- バンドの一番濃かった左から4番目のものを3mLのLB培地(amp+)で37℃で振とう培養した(overnight)
- pSB1A2(J22005)は左から6番目と7番目がバンドが見えたので、
- 7番目のものを3mLのLB培地(amp+)で37℃で振とう培養した(overnight)
- RFP(I13507)はバンドが現れなかった
- (3) PCR処理を行った4つ全てについてSodAが十分量含まれていることが27日(金)の電気泳動によりわかった
 "
"