Team:Kyoto/Notebook
From 2010.igem.org
(→Thursday, July 22 By: Wataru) |
(→Electrophoresis of the PCR products for 40min) |
||
| Line 82: | Line 82: | ||
====[[Team:Kyoto/Protocols#Electrophoresis|Electrophoresis]] of the PCR products for 40min==== | ====[[Team:Kyoto/Protocols#Electrophoresis|Electrophoresis]] of the PCR products for 40min==== | ||
[[Image:KyotoExp100722-1.png|300px|right]] | [[Image:KyotoExp100722-1.png|300px|right]] | ||
| - | + | Marker: 100bp, 1kb, 1kb, 100bp | |
| - | + | {| class="electrophoresis" | |
| - | + | !Lane||Name||Length(bp)||Result | |
| + | |- | ||
| + | |1||SRRz/Rz1||1386||⎼: | ||
| + | |- | ||
| + | |2||SRRz/Rz1||1386||⎼: | ||
| + | |- | ||
| + | |3||S||392||⎼: | ||
| + | |- | ||
| + | |4||S||392||⎼: | ||
| + | |- | ||
| + | |5||SRRz/Rz1||1386||⎼: | ||
| + | |- | ||
| + | |6||SRRz/Rz1||1386||⎼: | ||
| + | |- | ||
| + | |7||S||392||⎼: | ||
| + | |- | ||
| + | |8||S||392||⎼: | ||
</div> | </div> | ||
Revision as of 12:33, 6 October 2010
Index
Notebook
Tuesday, July 20 By: Wataru, Tomo, Yuki, Kazuya, Ken, Makoto
Transformation
| Name | Well | Sample (µl) | Competent Cells (µl) | Total (µl) | Plate | Incubation | Result |
|---|---|---|---|---|---|---|---|
| <partinfo>J23100</partinfo> | 1-18-C | 1 | 20 | 21 | LB (Amp+) | At 37℃, 7/20 20:50 - 7/21 17:00 | ○ |
| <partinfo>J23105</partinfo> | 1-18-M | 1 | 20 | 21 | ○ | ||
| <partinfo>J23116</partinfo> | 1-20-M | 1 | 20 | 21 | ○ | ||
| <partinfo>R0011</partinfo> | 1-6-G | 1 | 20 | 21 | ○ | ||
| <partinfo>E0840</partinfo> | 1-12-O | 1 | 20 | 21 | ○ | ||
| <partinfo>J06702</partinfo> | 2-8-E | 1 | 20 | 21 | ○ | ||
| <partinfo>pSB4K5</partinfo> | 1-5-G | 1 | 20 | 21 | × | ||
| <partinfo>B0015</partinfo> | 1-23-L | 1 | 20 | 21 | LB (Kan+) | × |
A vector of <partinfo>pSB4K5</partinfo> is Kanamycin-resistance, however, we plated it to LB plate (Amp+). And We didn't pre-culture <partinfo>B0015</partinfo> despite its vector is Kanamycin-resistance. So, it was predicted that we will fail the transformation of <partinfo>pSB4K5</partinfo> and <partinfo>B0015</partinfo>.
Wednesday, July 21 By: Wataru, Ken, Makoto, Takuya Y.
Culture at 37℃ from 07/21 20:50 to 07/22 17:00 and Making Master Plate
Transformation
| Name | Well | Sample (µl) | Competent Cells (µl) | Total (µl) | Plate | Incubation | Result |
|---|---|---|---|---|---|---|---|
| <partinfo>pSB4K5</partinfo> | 1-5-G | 1 | 20 | 21 | LB (Kan+) | At 37℃ 7/21 20:50 - 7/22 16:30 | ○ |
| <partinfo>B0015</partinfo> | 1-23-L | 1 | 20 | 21 | ○ |
PCR for SRRz/Rz1 and S
| No. | Water (µl) | MgSO4 | dNTPs | 10xBuffer | Template DNA | Primer Forward | Primer SRRz/Rz1 Reverse | Primer S Reverse | KOD Plus ver.2 | Total |
|---|---|---|---|---|---|---|---|---|---|---|
| 1 | 28 | 3 | 5 | 5 | 5 | 1.5 | 1.5 | - | 1 | 50 |
| 2 | 28 | 3 | 5 | 5 | 5 | 1.5 | 1.5 | - | 1 | 50 |
| 3 | 28 | 3 | 5 | 5 | 5 | 1.5 | - | 1.5 | 1 | 50 |
| 4 | 28 | 3 | 5 | 5 | 5 | 1.5 | - | 1.5 | 1 | 50 |
| 5 | 28 | 3 | 5 | 5 | 5 | 1.5 | 1.5 | - | 1 | 50 |
| 6 | 28 | 3 | 5 | 5 | 5 | 1.5 | 1.5 | - | 1 | 50 |
| 7 | 28 | 3 | 5 | 5 | 5 | 1.5 | - | 1.5 | 1 | 50 |
| 8 | 28 | 3 | 5 | 5 | 5 | 1.5 | - | 1.5 | 1 | 50 |
| 94℃ | 2min | |
| 98℃ | 10sec | 30 cycles |
| 55℃ | 30sec | |
| 68℃ | 4min | |
| 4℃ | forever |
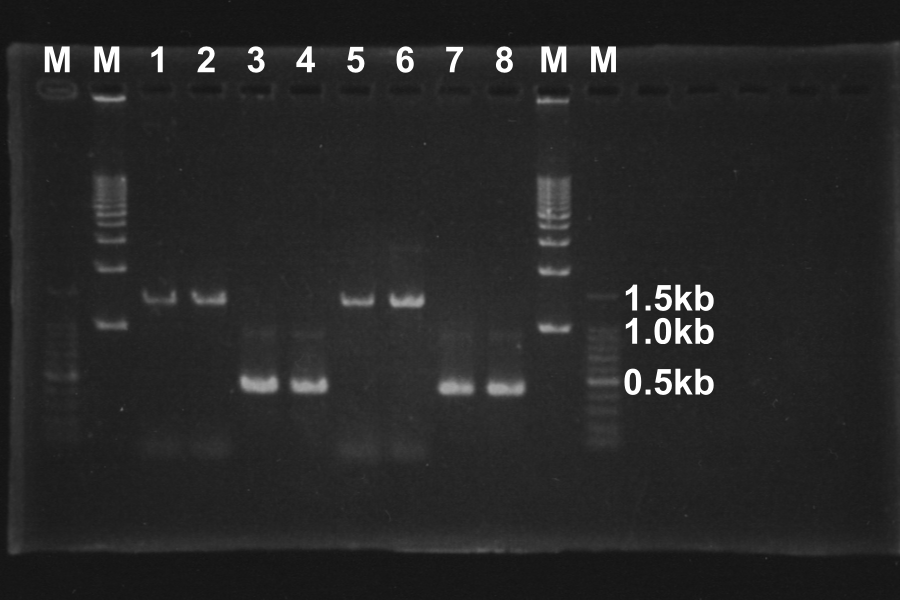
Thursday, July 22 By: Wataru
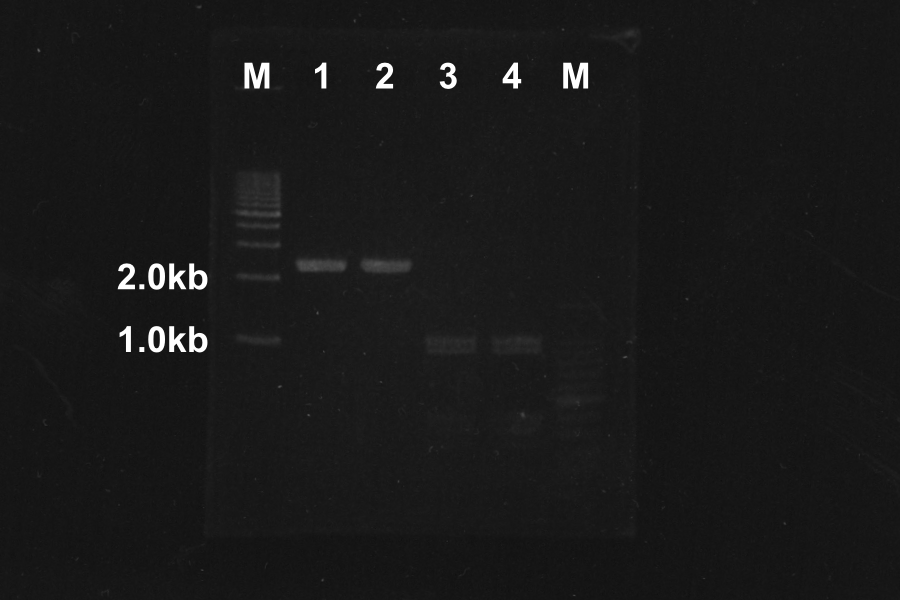
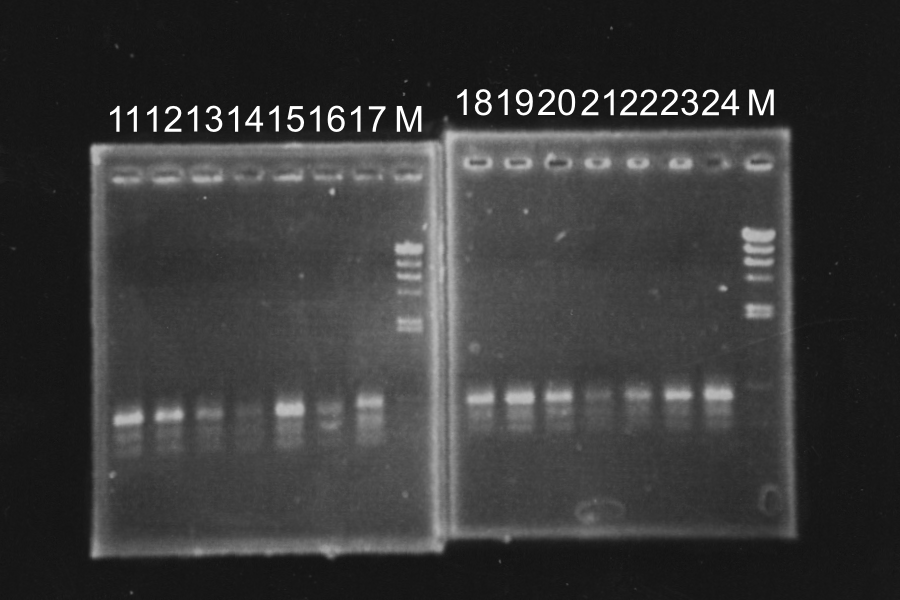
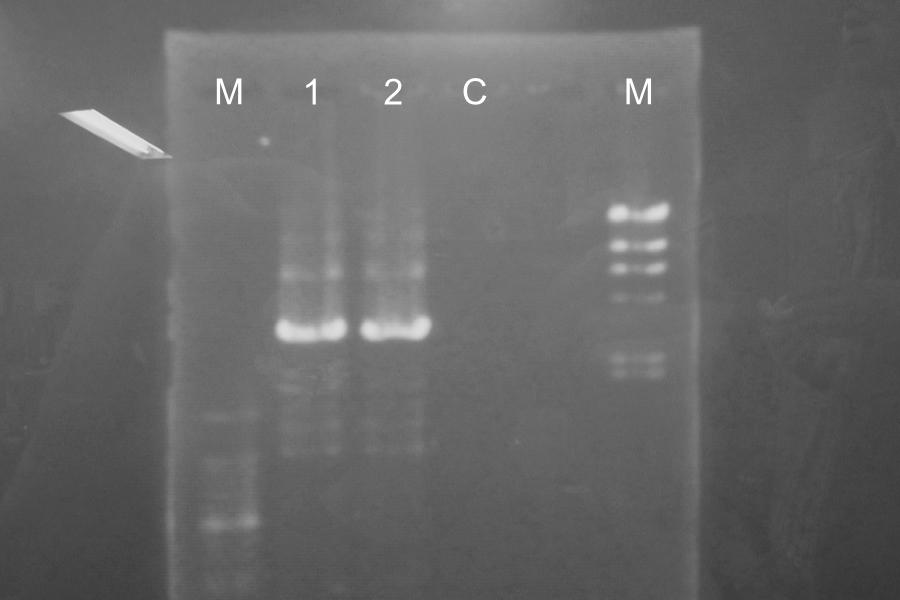
Electrophoresis of the PCR products for 40min
Marker: 100bp, 1kb, 1kb, 100bp
| Lane | Name | Length(bp) | Result | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 1 | SRRz/Rz1 | 1386 | ⎼: | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 2 | SRRz/Rz1 | 1386 | ⎼: | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 3 | S | 392 | ⎼: | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 4 | S | 392 | ⎼: | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 5 | SRRz/Rz1 | 1386 | ⎼: | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 6 | SRRz/Rz1 | 1386 | ⎼: | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 7 | S | 392 | ⎼: | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 8 | S | 392 | ⎼:
Miniprep
The concentration of all samples was very week. Probably our shaking incubation was week. Culture from 07/22 17:00 to 07/23 10:00 and Making Master Plates of <partinfo>pSB4K5</partinfo> and <partinfo>B0015</partinfo>Friday, July 23 By: Wataru, Tomo, MakotoMiniprep
We lost <partinfo>B0015</partinfo> by our mistake. The concentration of <partinfo>pSB4K5</partinfo> is high, so this condition of shaking incubation is moderate. Picking up number 1, 3, 5, and 7 of the products of PCR, and PCR-purification
The concentration of sample number 1 and 5, the PCR products of S-R-Rz/Rz1, is week, so we desided to retry PCR. Retry of Standard PCR for S-R-Rz/Rz1
Restriction Digestion of <partinfo>J06702</partinfo> by EcoRI, XbaI, SpeI, and PstI to check function of our Restriction Enzyme
Electrophoresis of above sample for 35minComparison to sample 5(control, circular DNA), the bands of sample 1, 2, 3, 4 was shifted. The DNA of sample 1, 2, 3, 4 was linearized by Restriction enzymes. So, our restriction enzymes work correctly. Digestion of the PCR products of S gene by EcoRI and SpeI and <partinfo>E0840</partinfo> by EcoRI and XbaI to insert S gene to <partinfo>E0840</partinfo>
After PCR purification, evaporated them and diluted 3ul. Ligation
Monday, July 26 By: Wataru, Tomonori, MakotoElectrophoresis of PCR ProductsAt the condition 4 (4.5µl MgSO4) and 6 (6µl MgSO4), S-R-Rz/Rz1 is amplified very much. So we decided to use them. PCR Purification
Transformation
Culture of <partinfo>pSB4K5</partinfo>, <partinfo>E0840</partinfo>, and <partinfo>B0015</partinfo>Tuesday, July 27 By: Wataru, Tomo, Kazuya, Ken, NaoiTeam:Kyoto/Protocols#Colony_PCR~Colony PCR of S-E0840 (Electrophoresis for 35min)
As a result, 1, 3, 5, 6, 11, 12, and 13 are inserted S gene correctly. So, we decided to use 6 as S-E08401 and 11 as S-E08402. Miniprep
Restriction Digestion
Transformation
Wednesday, July 28 By:Miniprep
Diluted S-E08401 and S-E08402 20 times with water, and used as template DNA. Deletion_PCR to delete a functional domain of S gene
Restriction Digestion to check the function of DpnI
Electrophoresis for 35min
DpnI works correctly.
Thursday, July 29 By:Restriction Digestion
Ligation and Pospholylation
Transformation
Monday, August 2 By: Wataru, KenMiniprep
Standard PCR of <partinfo>E0240</partinfo>E240 is very important parts to measure RPU of promoters in iGEM. However, we failed to transfect it to E.coli from parts kit of iGEM. So we decided to amplify this parts by PCR.
PCR Purification
Restriction Digestion for inserting <partinfo>E0240</partinfo> to pSB4K5 by 3A assembly
PCR Purification
Stored at -20℃. Error PCR
Transformation
Tuesday, August 3 By:Culture of each two colonies of SΔTMD1-E08401-1 and SΔTMD1-E08402 for 37℃ 08/03-08/04Miniprep for Construction of Measure(lacP) and Measure(Standard)
Restriction Digestion
PCR Purification
pSB4K5(E-P) is concentrated 10µL and E02401(X-P), E02402(X-P) are concentrated 1µL. Ethanol PrecipitationDilution of <partinfo>pSB4K5</partinfo> by 2µl MilliQLigation
Standard PCR of <partinfo>J23101</partinfo>-<partinfo>E0240</partinfo> that is important in the measurement of RPU
PCR Purification
Restriction Digestion
PCR Purification
J23101-E0240(E-P) is concentrated 7µL Ligation
Transformation
Thursday, August 5 By:Result of Transformation
pSB4K5 is inserted RFP generator. We didn't distinguish this inserted parts from low copy plasmid backbone, so self-ligated colony is red. So, white colony is correctly inserted parts. However, white colonies and green colonies are observed in R0011-E02401[Low] and R0011-E02402[Low] plate. We cultured both white and green colonies. In J23101-E0240[Low], Many of colonies are red, but green colonies are observed. We cultured green colonies.
As a result, deletion is succeeded, however, point mutation is failed. It is because DpnI is too little to digest all of template DNA.
Friday, August 6Miniprep
Restriction Digestion
Electrophoresis
Error PCR (Retry)
Transformation
Monday, August 9 By: Wataru, Tomonori, Ken, TakuyaMiniprep of MS and ML
Transfotrmation of MS and ML
Restriction enzyme digestion and ethanol precipitationTo use lac p for next ligation, we digested 1-6-G by EroRI and PstI
Incubate 37℃ 8/9 16:20‾18:20 After restriction enzyme digestion, we did ethanol precipitation. Ligation and Transformation
Making culture plate on lac p (low), MS and ML
Minprep of ΔTMD1+GFP
37℃ 8/9 18:00‾8/10 9:00 Culture and Master Plate
Discussion: About sample 1, 3, 4, 5 and 7, lac promoter was correctly inserted in low copy plasmid. About sample 2 and 6, low copy plasmid and vector derived from lac promoter were ligated. We decided to use sample 1 or 3. Miniprep of C2+lac(low), S-R-Rz 1', 3'lac(low)1 : 31.2 (ng/µL) lac(low)2 : 29.9 (ng/µL) Restriction Digestion and electrophoresis of lac (low) 1 and 3
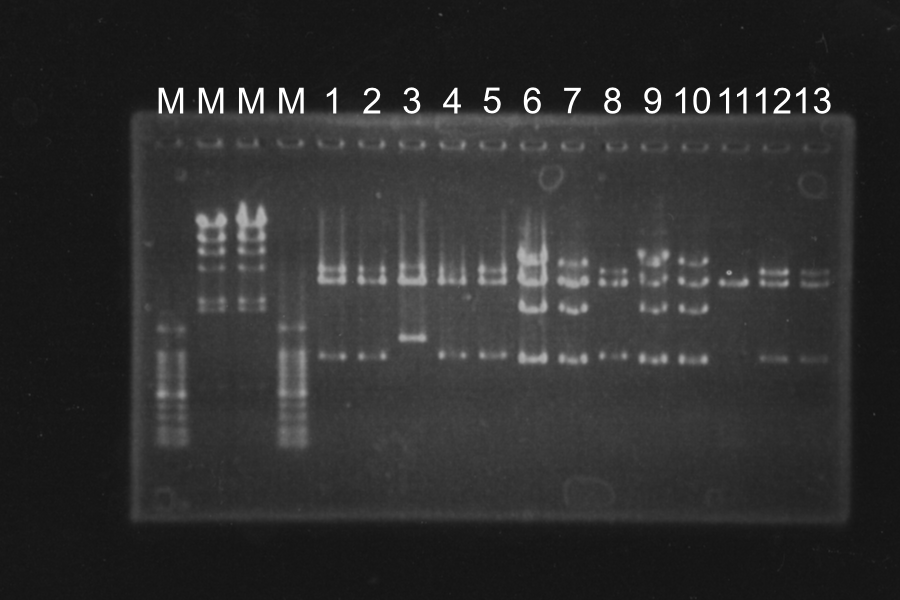
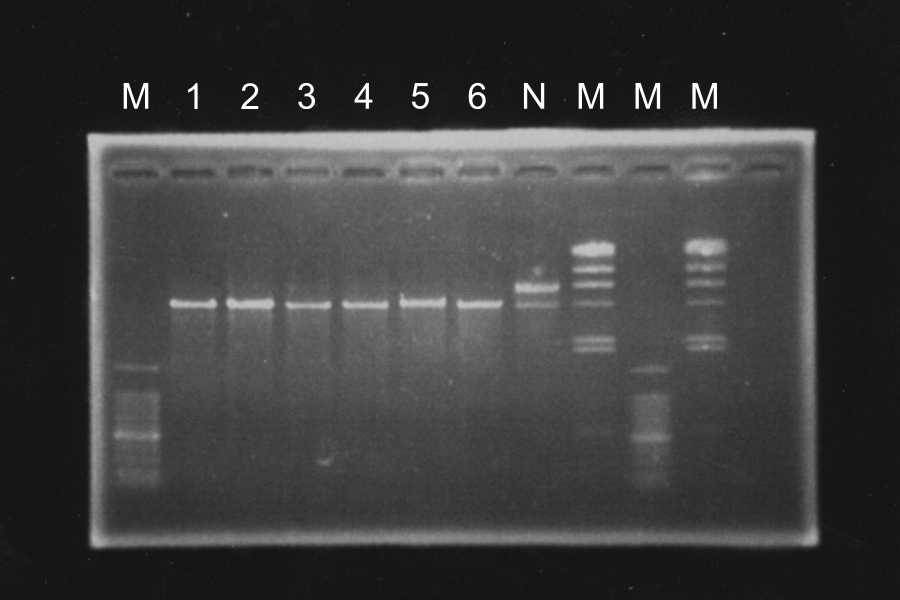
Sample: 1-1, 1-2, 1-3, 1-N, 3-1, 3-2, 3-3, 3-N M 1-1 1-2 1-3 1-N M M 3-1 3-2 3-3 3-N M Discussion: Each enzyme correctly cut samples. Screening PCR of SRRzSample: 1-20 Control: P(1-23L) P'(2-8E) N Maker: lambda M N P P' P 1 2 3 4 5 6 M 7 8 9 10 11 12 13 M 14 15 16 18 19 20 M Discussion: All of the sample were self-ligation of DT and SRRz weren't inserted.
Thursday, August 12 By: Wataru, KenRestriction Digestion and electrophoresis of <partinfo>B0015</partinfo>
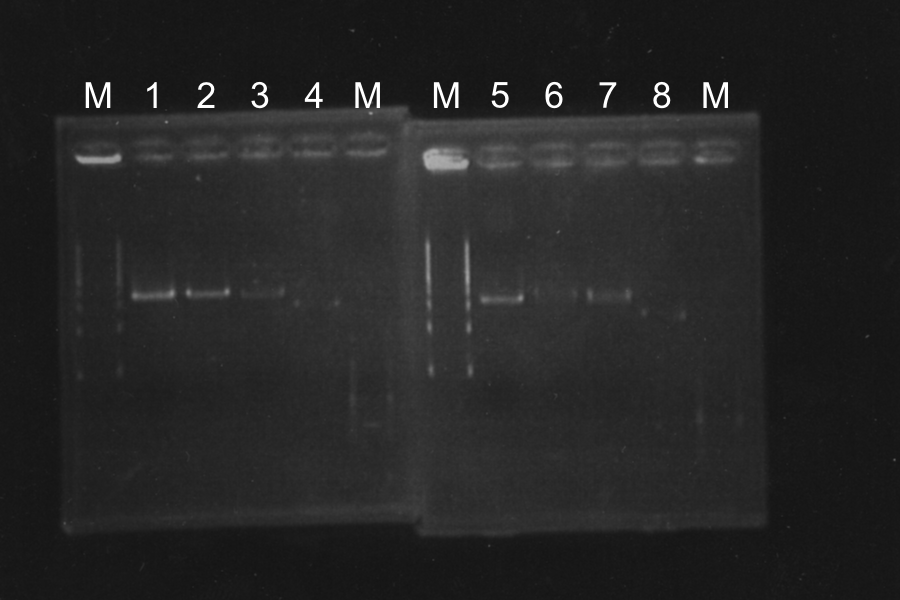
Sample: 1-6, N Maker: lambda, 100 M 1 2 3 4 5 6 N M M M Discussion: Each enzyme correctly cut each sample and was active.
Miniprep of SΔTMD1GFP29.6(ng/µg) Point mutation PCR of ΔTMD1GFP
Restriction Digestion(DpnI): 17:50-18:50Ligation and TransformationWe named point mutation PCR products rΔTMD1GFP.
Monday, August 23 By: Wataru, Tomo, Ken, Fumitaka, TasukuMiniprep of ΔTMD1
Sequencing of ΔTMD1 and MSSample: rδTMD1GFP1-1, 2-2, and MS Discussion: The sequencing was in success and the results were desirable. It meant point mutation of δTMD1GFP was succeeded and sequence of MS was confirmed. We decided to use rδTMD1GFP. Screening PCR of SRRz-DTSample: 1-13, Marker: lambda and 100, Control:P(1-23L) and N
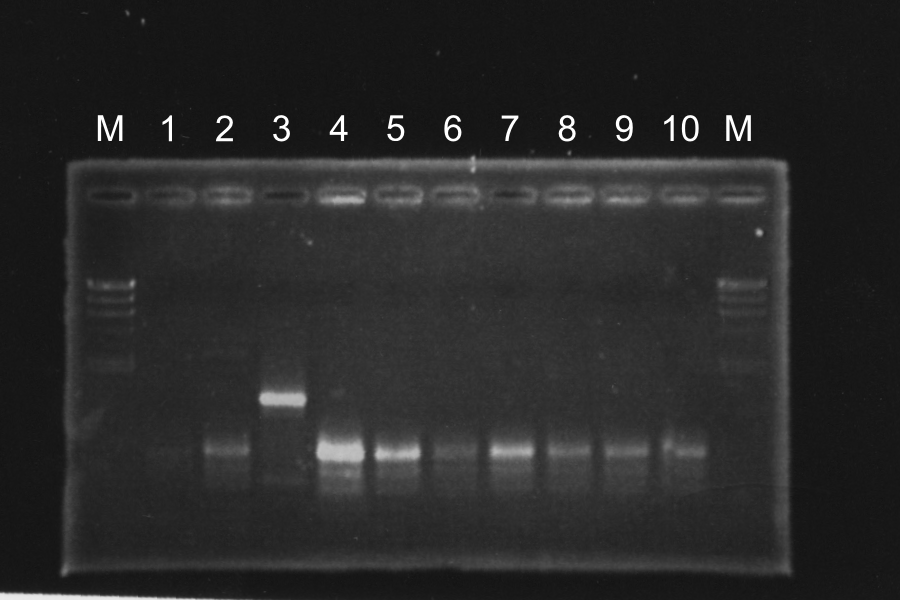
M 1 2 3 4 5 6 7 8 9 10 11 12 13 P N M Discussion: We found the band; about 200bp, and it meant the lligation was completed successfully. Deletion PCR of rΔTMD1GFP 2-2
Restriction Digestion(DpnI)
19:10-20:10 Ligation
20:15-21:15 TransformationWe named sample 1, 2 and control rrδTMD1GFP1, 2 and control.
Tuesday, August 24 By:Ken, Tomo, Tasuku, TakuyaRetry of deletion PCR of rδTMD1 GFP
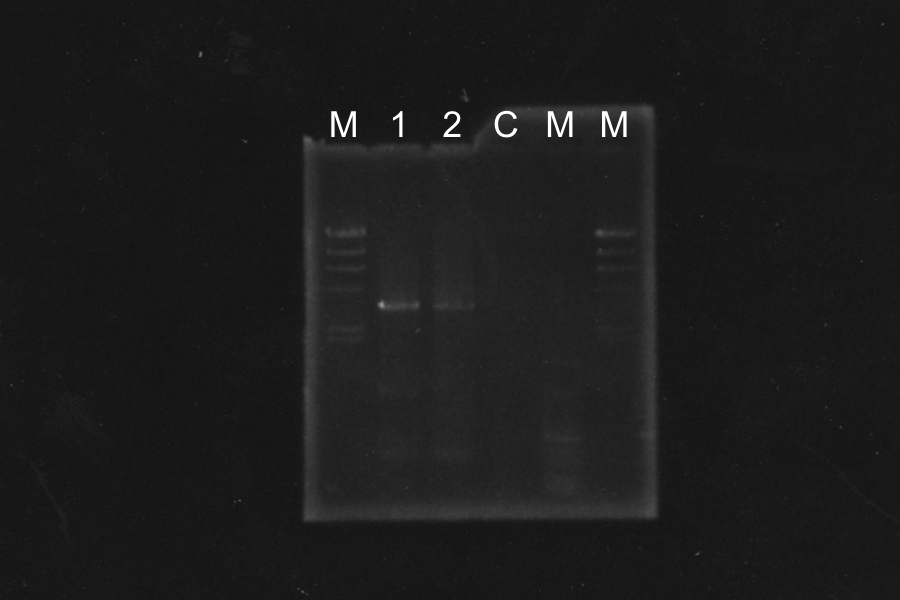
Restriction Digestion (DpnI)14:15-15:15 ElectrophoreisSample: 1, 2, and control, Maker: 100 and lambda M 1 2 C M We found the band of sample 1 and 2 about 3000bp and there wasn't the band of sample control. So, we confirmed the PCR and RE were completed successfully. LigationPoint mutation of SRRz
Restriction Digestion(DpnI), electrophoresis and ligationWe could find point mutation PCR and restriction enzyme of DpnI was done. =PCR of E0240
=PCR PurificationSample1: 5.5*50(ng/µL) Sample2: 5.2*50(ng/µL) Restriction Digestion(EcoRI, PstI) and Gel extractionSample1: 28.8 (ng/µL) Sample2: 26.4 (ng/µL) TransformationSample: rrΔTMD1GFP1. 2. control, and rSRRz1. 2. control
Wednesday, August 25 By:Ken, Tomo, Kazuya, Tasuku, TakuyaMaking culture and Master plate
Miniprep of 1-5G29.0 (ng/µL) Restriction Digestion and purification of 1-5G(low copy plasmid) and lac low
Ligation of <partinfo>E0240</partinfo> and <partinfo>pSB4K5</partinfo>, Transformation
Miniprep
Restriction Digestion of constP(0.7)
Purification of constP (0.7)49.8 ng/µL
Friday, August 27 By:Ken, Tomo, Kazuya, FumitakaMaking master plate of E0240 low
Restriction Digestion of rrΔTMD1 and rSRRz
(13:20-14:20) Purification
Lagation and transformationlacP + rrΔTMD1 1-2 constP (0.7) + rrΔTMD1 1-2 lac low + rSRRz 1-1
Monday, August 30 By: Tomonori, Kazuya, Tasuku, KenMaking culture and Master plate
Discussion: There ware some colonies, which emitted green light, on the plate 1. So, we cultured those colonies on master plate. On the plate 5 and 6, even though we used KRX, which is able to repress lac promoter, colonies might be dead. However, we still have to do some experience so that we confirm lac promoter cannot repress enough and E. coli cannot survive.
Miniprep
RE of constP (0.3) and lac rrΔTMD1Gel Extraction of lac rrΔTMD145min Discussion: There were two band at the bottom of the gel. It was too long -45min-, and insert and vector might be contaminated. But we went on next operation. Purification of constP (0.3) and lac rrΔTMD1
Ligation and transformation
Making culture and Master plate
Screenig PCR of lacP-rrΔTMD1GFP-constP Sample: 1-13 Control: Positive (1-23L) Maker: lambda, 100 M 1 2 3 4 5 6 7 8 9 10 11 12 13 P M Discussion: All of the sample except sample 10 might be self-ligation products of constP. Miniprep
Restriction Digestion of rSRRz and low
(13:25-14:30) Purification
Ligation and transformationInsert: rSRRz 1-1 Vector: low copy plasmid
Thursday, September 2 By: Tomonori, Tomo, Takuya, KenMaking culture and Master plate
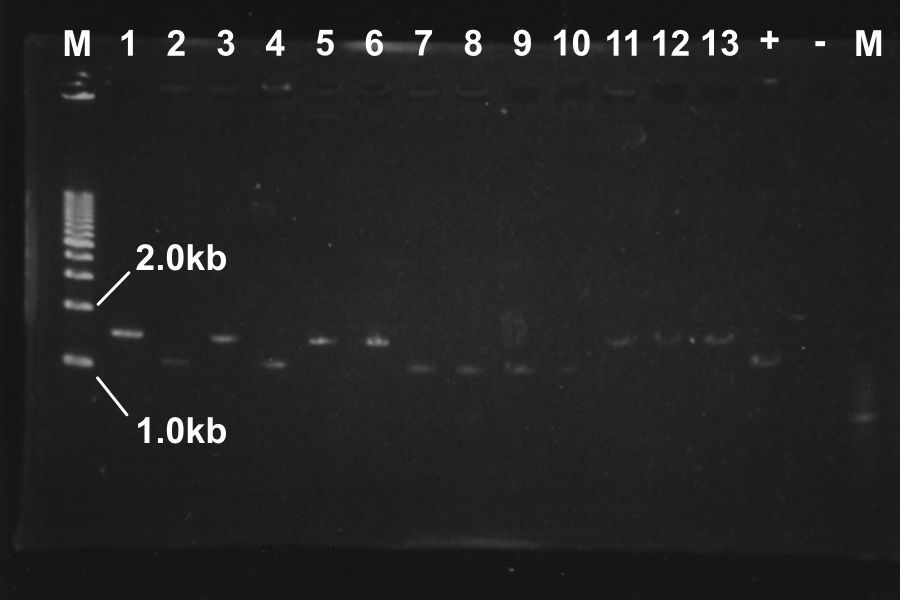
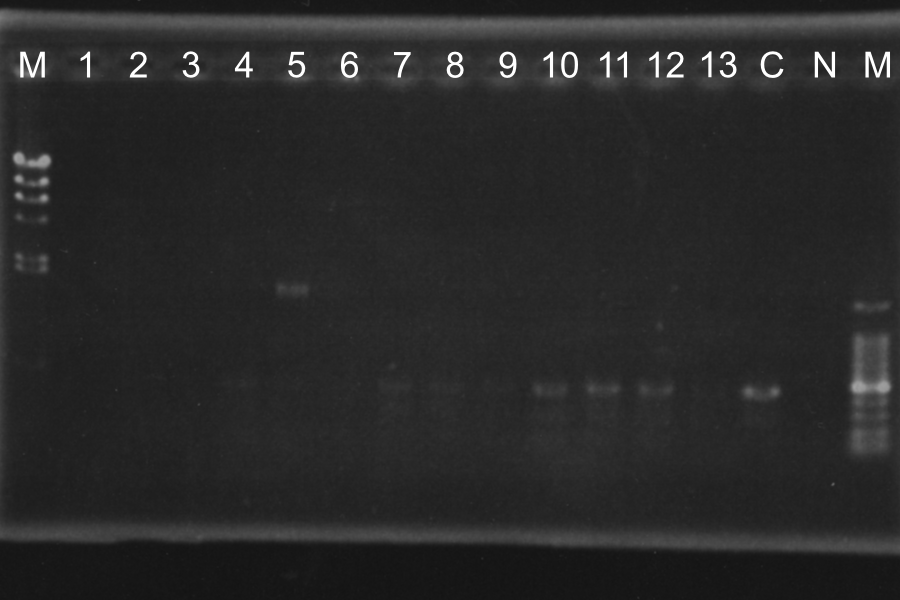
Screening PCR of rSRRz lowSample: rSRRz (1-13) Maker: lambda, 100 Control: Positive (1-23L), Neganive M 1 2 3 4 5 6 7 8 9 10 11 12 13 P N M Discussion: From sample 1, two vectors might be ligated. Sample 3 and 4, rSRRz might be inserted in low copy plasmid correctly. Sample 11, it might be the self-ligation product of low copy plasmid. Anyway, we decided to culture those 4 colonies on master plate. Friday, September 3 By: Tomonori, Tomo, Kazuya, Tasuku, Fumitaka, KenMaking culturelac rrΔTMD1 1, 3 rrΔTMD1 1-1, 1-2 rSRRz 1-1, 1-2 ML
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
 "
"