Team:LMU-Munich/Cut'N'survive/Schedule
From 2010.igem.org
(→Aim 1: DNA replication+PCR) |
|||
| Line 48: | Line 48: | ||
Primer used:[[Team:LMU-Munich/Cut'N'survive/Schedule/Primer13| 13TrecF]][[Team:LMU-Munich/Cut'N'survive/Schedule/Primer14| 14TrecMutSR]] | Primer used:[[Team:LMU-Munich/Cut'N'survive/Schedule/Primer13| 13TrecF]][[Team:LMU-Munich/Cut'N'survive/Schedule/Primer14| 14TrecMutSR]] | ||
| - | PCR7b: replication of the p14*TEVrecogn with mutagenisis (Spe1) [ ] | + | PCR7b: replication of the p14*TEVrecogn with mutagenisis (Spe1) with TEV recogn in primer (16TrecR) [ ] |
[[Image:PCR7b.jpg]] | [[Image:PCR7b.jpg]] | ||
Primer used:[[Team:LMU-Munich/Cut'N'survive/Schedule/Primer15| 15TrecMutSF]][[Team:LMU-Munich/Cut'N'survive/Schedule/Primer16| 16TrecR]] | Primer used:[[Team:LMU-Munich/Cut'N'survive/Schedule/Primer15| 15TrecMutSF]][[Team:LMU-Munich/Cut'N'survive/Schedule/Primer16| 16TrecR]] | ||
| - | PCR8: joining PCR of p14*TEVrecogn [ ] | + | PCR8: joining PCR of p14*TEVrecogn with TEV recogn in primer (16TrecR) [ ] |
[[Image:PCR8.jpg]] | [[Image:PCR8.jpg]] | ||
Revision as of 13:23, 18 August 2010
PCR1: replication of the Tet-inducible CMV promotor [X]
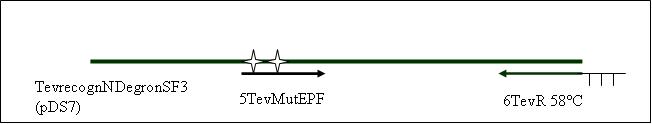
PCR2a: replication of the TEVrecogn-NDegron-SF3 Part with mutagenisis (EcoR1 + Pst1) [ ]
Primer used: 3TevF 4TevMutEPR
PCR2b: replication of the TEVrecogn-NDegron-SF3 Part with mutagenisis (EcoR1 + Pst1) [ ]
Primer used: 5TevMutEPF 6TevR
PCR3: joining PCR of the TEVrecogn-NDegron-SF3 Part [ ]
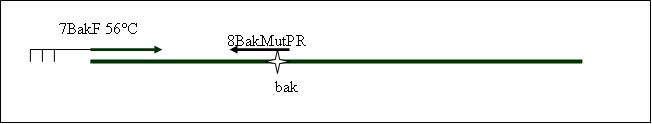
PCR4a: replication human bak with mutagenisis (Pst1) [ ]
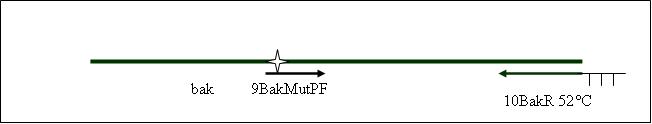
PCR4b: replication human bak with mutagenisis (Pst1) [ ]
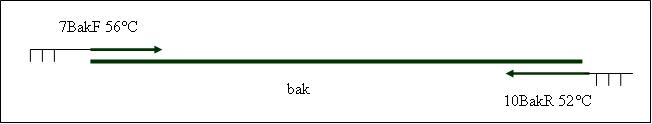
PCR5: joining PCR of human bak [ ]
PCR6: replication of the SV40-polyadenylation site [X]
PCR7a: replication of the p14*TEVrecogn with mutagenisis (Spe1) [ ]
Primer used: 13TrecF 14TrecMutSR
PCR7b: replication of the p14*TEVrecogn with mutagenisis (Spe1) with TEV recogn in primer (16TrecR) [ ]
Primer used: 15TrecMutSF 16TrecR
PCR8: joining PCR of p14*TEVrecogn with TEV recogn in primer (16TrecR) [ ]
oder
alt:
Meilensteine
Für die Projekte haben wir uns jeweils folgende „Meilensteine“ gesetzt:
„TEV-System“ 1. Verknüpfen von Bak mit interagierendem Protein A, einem N-Degron und einer Schnittstelle für die TEV-Protesase, sodass Bak noch aktiv bleibt. Davor ist ein induzierbarer Promotor und eine Antibiotikaresistenz geschaltet (Nachweis: induzierbarer Zelltod) Aufwand: ca. 6-8 Wochen, 2000€
2. Verknüpfen von eGFP als Zielgen mit einer TEV-Schnittstelle, einer TEV-Protease, und interagierendem Protein B. (Nachweis: eGFP leuchtet, Proteinchromatographie)
Aufwand: ca. 6-8 Wochen, 2000€, Parallele Arbeit ist möglich und notwendig
3. Einbringen beider Konstrukte in Zellen (Nachweis: eGFP leuchtet, einige Zellen sterben, Proteinchromatographie)
Aufwand: ca. 2 Wochen, 1000€
4. Parallel wird versucht, das erste Konstrukt stabil ins Zellgenom einzubauen.
Aufwand: ca. 2 Wochen, 500€


![]()
![]()







![]()
![]()

![]()
Contents
Aim 1: DNA replication+PCR






Aim 2: Assembling Biobricks
Aim 3: Testing products
Aim 4: Testing system
![]()
![]()

 "
"