Team:UT-Tokyo/Sudoku lab note
From 2010.igem.org
m (→Terminator leak switch) |
m |
||
| Line 193: | Line 193: | ||
[[Image:Sudoku_Hin_making.png|200px|thumb|How to make Hin parts]] | [[Image:Sudoku_Hin_making.png|200px|thumb|How to make Hin parts]] | ||
| - | We | + | We get Hin parts from HQ (BBa_J31000). |
| + | |||
| + | We use this part as the form of reverse, so we made this part reverse by PCR. | ||
| + | |||
===Expression check=== | ===Expression check=== | ||
Revision as of 00:33, 16 October 2010


Sudoku
Abstract Construct Lab note [ June / July / August ] Result
Lab note
To make main construct, we made several assays.
Detail protocols about this is:
June.
July.
Main construct
If you want to know detail explanation, please read Construct section.
Assay
Terminator leak switch
To realize 4C3 leak switch, we should choose proper terminator which terminates transcription when connected two or more but leak when single.
We made "A-M-RO/Char" assay (see Fig, named as the famous animation, "MOBILE SUIT GUNDAM") to select proper terminator:
AOI
no terminator
-> cre protein express rapidly
-> lox site is removed rapidly
-> gfp may expression rapidly
MURASAKI
one terminator
-> cre protein express slowly
-> lox site is removed slowly
-> gfp may expression slowly
ROKUJYO
two terminator
-> cre protein can't express
-> lox site remain
-> gfp expression may not express
First we use single terminator BBa_B1006.
Then other terminators are tested: 80%-terminate terminator and 99%-terminate terminator to determine the best threshold to realize terminator leak switch.
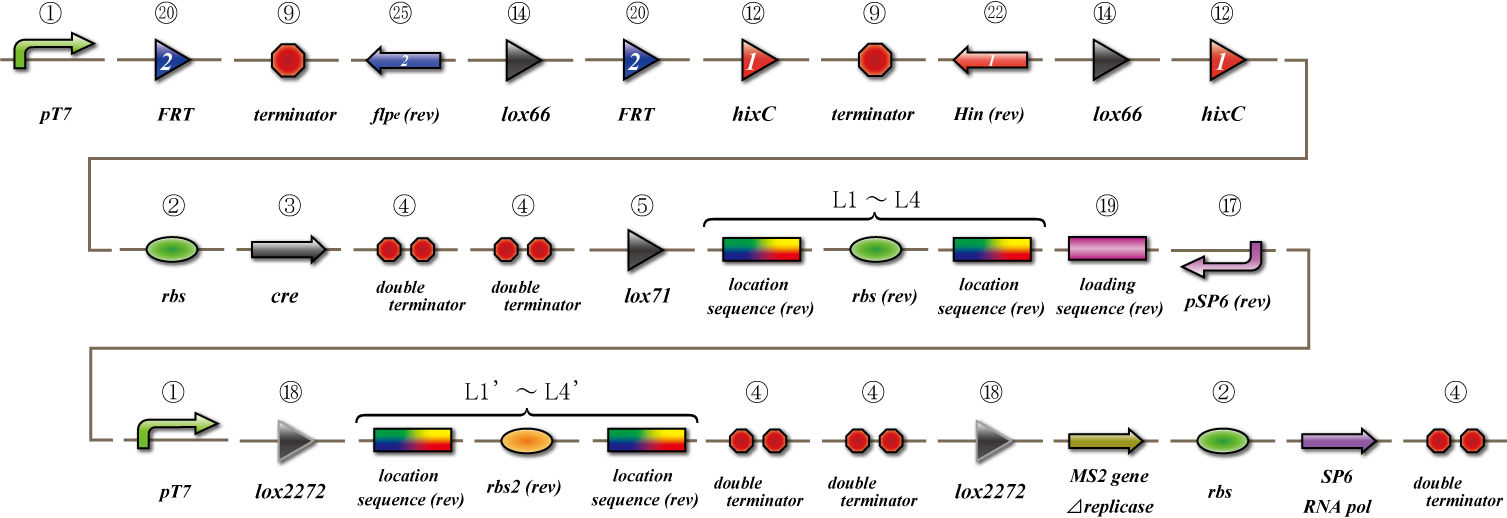
Location sequence
Parts making
Ligate small parts - recognation site, location site and rbs.
Expression check
location sequence を用いた、mRNA の相補鎖による翻訳の抑制を調べるassay。アンチセンス鎖 として働くL1'-L4' をc-pro を用いて転写。c-pro は恒常的に作動するプロモーター。iGEM パー ツに存在するconstitutive promoter で最も強いものを使用。pBAD(rev) はアラビノースを投入する ことで作動するプロモーター。アラビノースの濃度と転写量の関係は不明。そこで、まずは各プロ モーターの転写を開始させる強さを調べるアッセイを行う。それぞれのプロモーター(pBAD は正方 向のものを用いる) の下流にgfp を発現するparts をつなげることで、gfp をレポートタンパク質と して用いて、転写の強さを調べる。 対照実験として、ポジティブコントロールを作成。アンチセンス鎖(L1'-L4') が存在しないなかで、 L1-L4&loading sequence の配列を通過してtet 耐性が見られるかどうかを確認。 として、現在設計されているlocation sequence に開始コドンに対応する塩基配列が含まれ ているものを発見(L1,L4)。rbs の後ろに配列があるため、gfp に余分なアミノ酸が結合するか、も しくは、完全に配列が変化するかして、いい実験結果が得られない可能性が高いため、assay 系は、 L2,L4 のみを用いて行う。
MS2 virus
Parts making
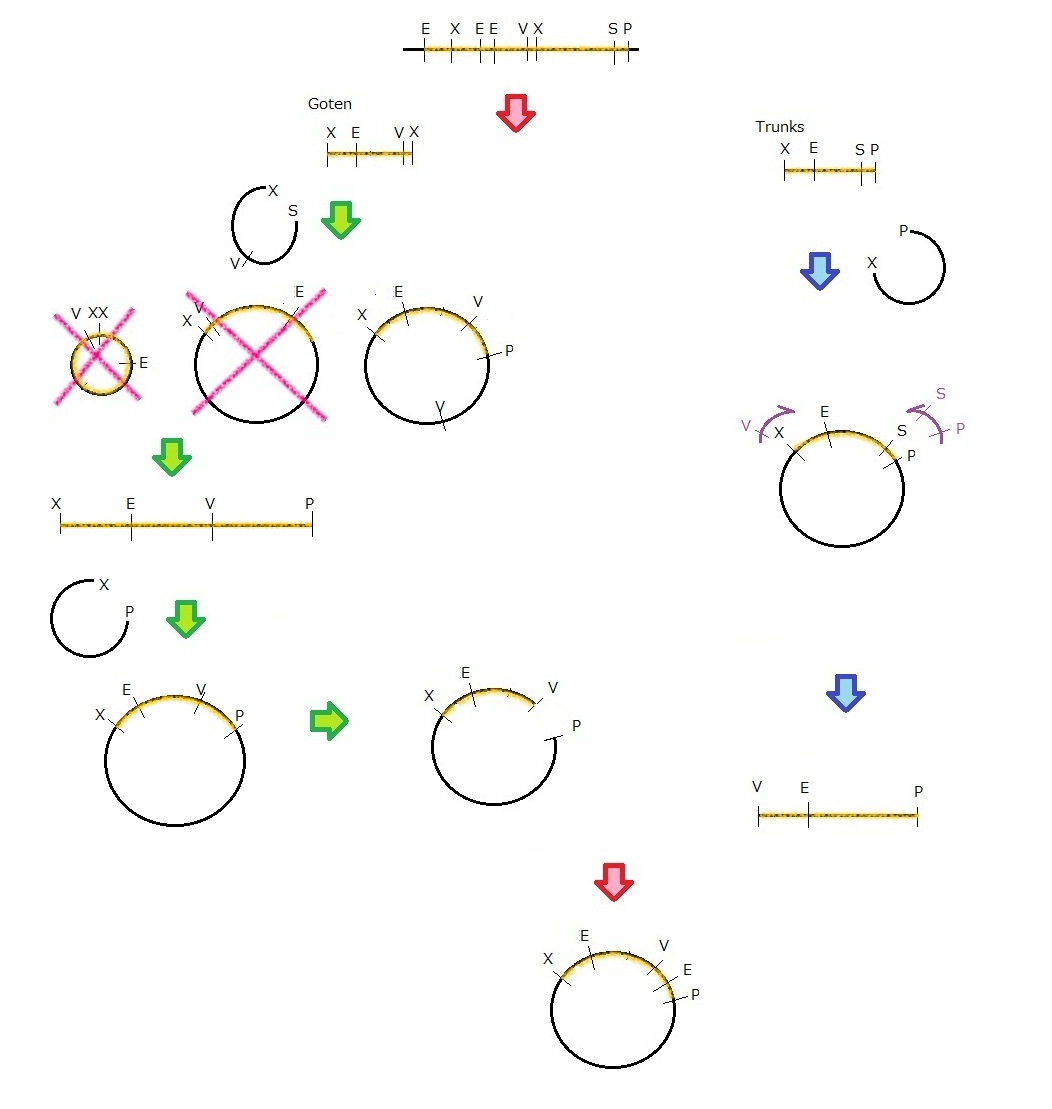
We get MS2 gene RT-PCR product. This original product include a lot of restricted enzyme site: two EcoRI site, two XbaI site, one SpeI site and one VspI site. To make this DNA the iGEM part, PCR making mutation is done.
1. MS2 のままでは長すぎて、操作しにくいため、pre�x, suffix を用いて、PCR で増幅した パーツをXP cut。XX で挟まれている側をGoten,XP で挟まれている側をTranks とする。 2. Goten をXScut されたamp 耐性空ベクターとライゲーション。セルフライゲーション、 insert が逆向きに入ったもの、正向きに入ったものの3 通りのつながり方が考えられる が、セルフライゲーションしたプラスミドにはクロラムフェニコール耐性がないため、ク ロラムフェニコールプレートで選択的に排除される。目的と逆向きにinsert とvector が ligation してしまった場合は、次の制限酵素処理したものを電気泳動する際に目的のパー ツと分離することができる。 3. Tranks はXPcut されたvector とligation されたのち、X サイトを改変し、かつ、V サ イトを新たに加えるように設計されたプライマーを用いて、PCR によりDNA 鎖を増幅 する。 4. Goten を挿入したplasmid をVPcut したのち、PCR によって増幅されたTranks のVPcut 産物およびXPcut された空ベクターと3 つ同時にligation。
1. MS2 のままでは長すぎて、操作しにくいため、pre�x, suffix を用いて、PCR で増幅した パーツをXP cut。XX で挟まれている側をGoten,XP で挟まれている側をTranks とする。 2. Goten は初め、amp 耐性のベクターとligation。セルフライゲーション、insert が逆向き に入ったもの、正向きに入ったものの3 通りのつながり方が考えられるが、セルフライ ゲーションしたinsert パーツにはクロラムフェニコール耐性がないため、クロラムフェニ コールプレートで選択的に排除される。目的と逆向きにinsert とvector がligation して しまった場合は、次の制限酵素処理したものを電気泳動する際に目的のパーツと分離する ことができる。E サイトを改変しようとしたが、失敗したため、とりあえず、VPcut し て、改変したTranks とつなげようとした。しかし、amp 耐性ベクターはベプター部位に もV サイトを持っているため、一度XPcut して、CP 耐性のベクターに載せ替えたのち、 VPcut して、改変したTranks とligation. 3. Tranks はXPcut されたvector とligation されたのち、X サイトを改変し、かつ、V サ イトを新たに加えるように設計されたプライマーを用いて、PCR によりDNA 鎖を増幅 する。 4. Goten を挿入したplasmid をVPcut したのち、PCR によって増幅されたTranks のVPcut 産物とligation。
Expression check
We use MS2 phage to transmit information of location and number. The expression of the phage should start after 4C3 leak switch turns on. MS2 phage transport RNA which has loding sequence, so we knock out self assembly and made our E.coli translate loading seaquence at another point. To check whether transportion go correctly, we made assay.
flpe
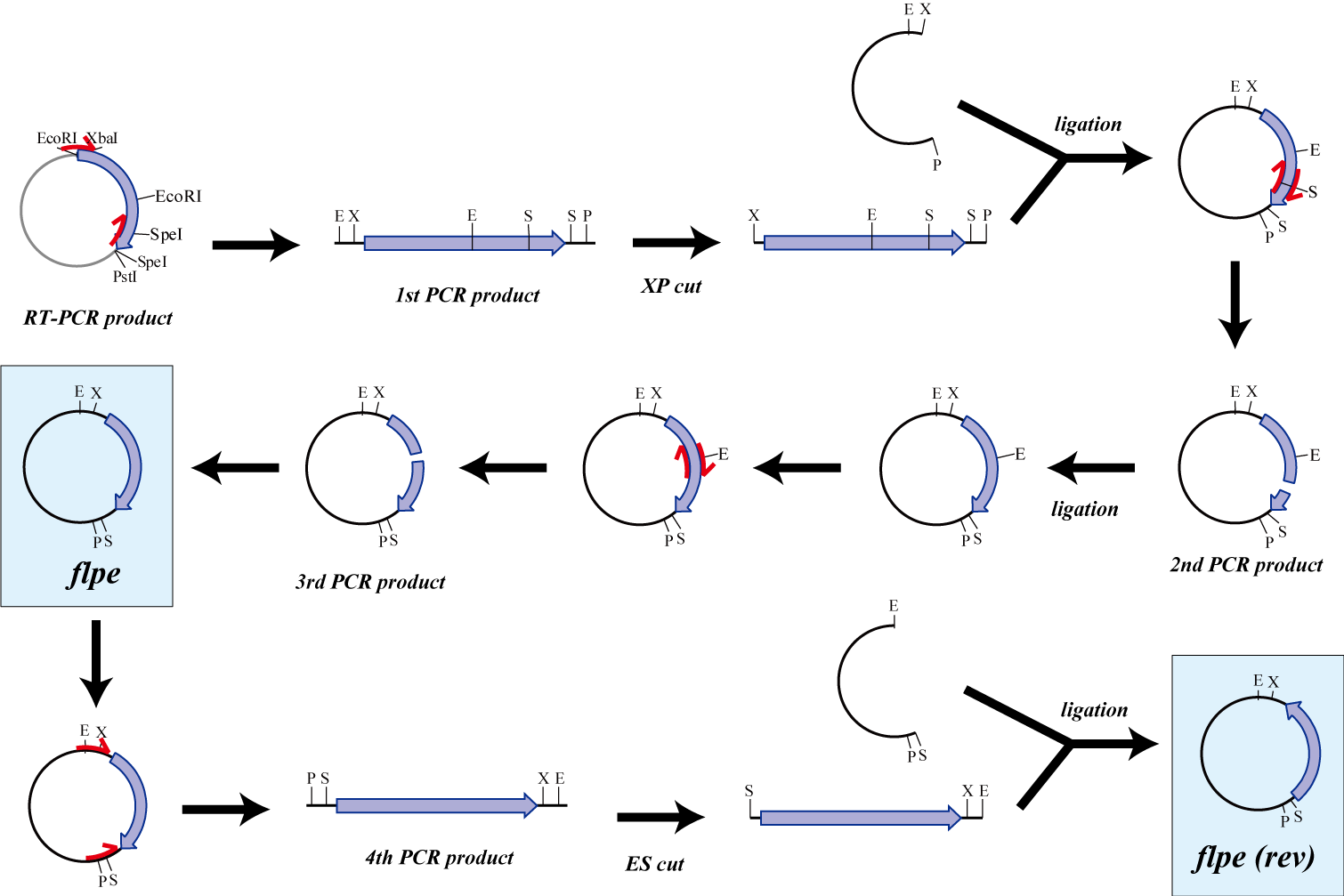
Parts making
The original flpe include two restriction enzyme sites(EcoRI, SpeI).
We modified this flpe by PCR:
1st PCR : cloning
-> ligation with vector
-> 2nd PCR : modified SpeI site
-> 3rd PCR : modified EcoRI site
We use this part as the form of reverse, so we made this part reverse by PCR.
Expression check
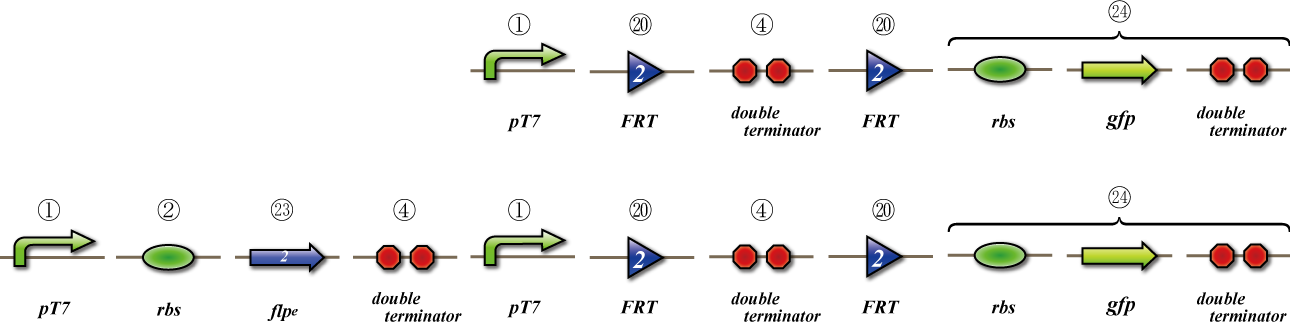
To check whether flpe works correctly, we made assay shown in Fig.
The top construct is a nagative control. GFP can't be expressed because of the double terminators.
The bottom construct express GFP when flpe works correctly. When flpe is expressed correctly, flpe recognaize the frt site and double terminator which terminates the expression of gfp is removed, so GFP may be expressed.
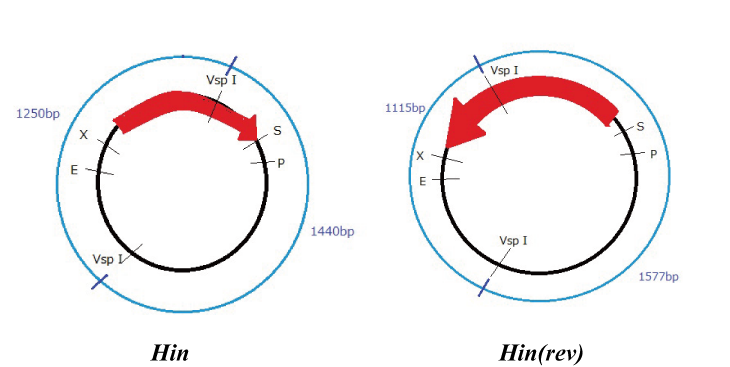
Hin
Parts making (reverse)
We get Hin parts from HQ (BBa_J31000).
We use this part as the form of reverse, so we made this part reverse by PCR.
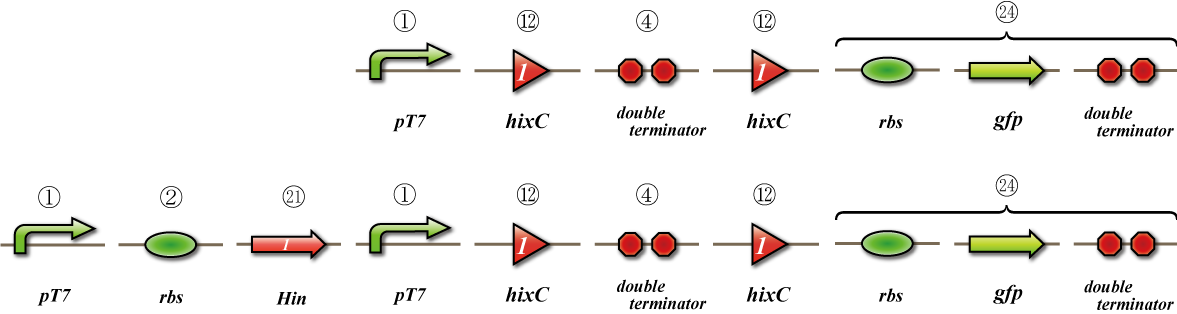
Expression check
To check whether flpe works correctly, we made assay shown in Fig, similar to the assay of flpe check.
The top construct is a nagative control. GFP can't be expressed because of the double terminators.
The bottom construct express GFP when Hin works correctly. When Hin is expressed correctly, Hin recognaize the hix site and double terminator which terminates the expression of gfp is removed, so GFP may be expressed.
Parts list
| Number | Name | Link to BioBrick | Plate coordinate | Vector | Code length |
| 1 | T7 promoter | plate1-6N | pSB1AK8 | 46bp | |
| 2 | rbs | plate1-1H | pSB1A2 | 15bp | |
| 3 | cre recombinase | plate1-5D | pSB1A2 | 1037bp | |
| 4 | double terminator | plate2-24C | pSB1AK3 | 95bp | |
| 5 | lox66 recombinase site | plate1-17J | pSB1A2 | 34bp | |
| 6 | Kan resistance (rev) | plate1-2K | pSB1A2 | 816bp | |
| 7 | rbs (rev) | plate1-1J | pSB1A2 | 15bp | |
| 8 | lox71 recombinase site |
order-made | --- | --- | 34bp |
| 9 | single terminator | plate1-4H | pSB1AK3 | 39bp | |
| 10 | constant express promoter | plate1-18A | pSB1A2 | 35bp | |
| 11 | Tet resistance (rev) | plate1-1N | pSB1A2 | 1191bp | |
| 12 | hixC | plate1-1B | pSB1A2 | 26bp | |
| 13 | --- |
--- | --- | --- | --- |
| 14 | lox66 | plate1-17H | pSB1A2 | 34bp | |
| 15 | rbs-mRFP1-terminator | plate1-22O | pSB1A2 | 861bp | |
| 16 | location sequence(old) |
order | --- | --- | --- |
| 17 | pSP6(rev) |
order | --- | --- | --- |
| 18 | lox2272 |
order | --- | --- | --- |
| 19 | loading sequence(rev) |
order | --- | --- | --- |
| 20 | frt | From HQ | pSB1A2 | 34bp | |
| 21 | hin(true) | From HQ | ??? | 573bp | |
| 22 | hin(rev) |
--- | --- | ??? | 573bp |
| 23 | flpe |
--- | --- | --- | about 1.2kbp |
| 24 | gfp unit |
--- | --- | --- | about 1.2kbp |
| 25 | flpe(rev) |
--- | --- | --- | about 1.2kbp |
 "
"